Nat Commun:帕金森疾病背后的肠道秘密,科学家揭示微生物组的“肠脑轴”作用
时间:2025-06-16
来源:100医药网 2025-06-16 16:58
本文通过大规模的元分析揭示了帕金森病患者肠道微生物组的显著变化,并评估了其在疾病诊断中的潜在应用价值。帕金森疾病(PD,Parkinson s disease)是仅次于的第二大神经退行性疾病,其发病率在全球范围内呈上升趋势。据估计,截至到2050年全球帕金森疾病患者的数量可能会达到1200万,帕金森疾病的发病机制复杂,除了遗传因素外,诸如农药、溶剂等化学物质的暴露也被认为是重要的风险因素。
近年来,随着对研究的不断深入,科学家们发现肠道微生物组与帕金森疾病之间存在着密切的联系,而且越来越多的证据表明,肠道微生物组的改变可能与帕金森疾病的发生和发展有关,甚至可能通过 肠脑轴 影响系统的功能。然而,目前对于帕金森疾病相关肠道微生物组特征的研究结果并不一致,缺乏统一的。因此,全面了解帕金森疾病患者的肠道微生物组变化,并探索其在疾病和治疗中的潜在应用,已成为当前研究的热点和难点。
近日,一篇发表在国际杂志Nature Communications上题为 Machine learning-based meta-analysis reveals gut microbiome alterations associated with Parkinson s disease 的研究报告中,来自欧洲分子生物学实验室等机构的科学家们通过大规模的机器学习元分析,系统评估帕金森疾病患者肠道微生物组的变化并探索了其在疾病诊断中的潜在价值;研究人员旨在揭示帕金森疾病相关肠道微生物组的特征,并为开发基于肠道微生物组的诊断工具提供科学依据。
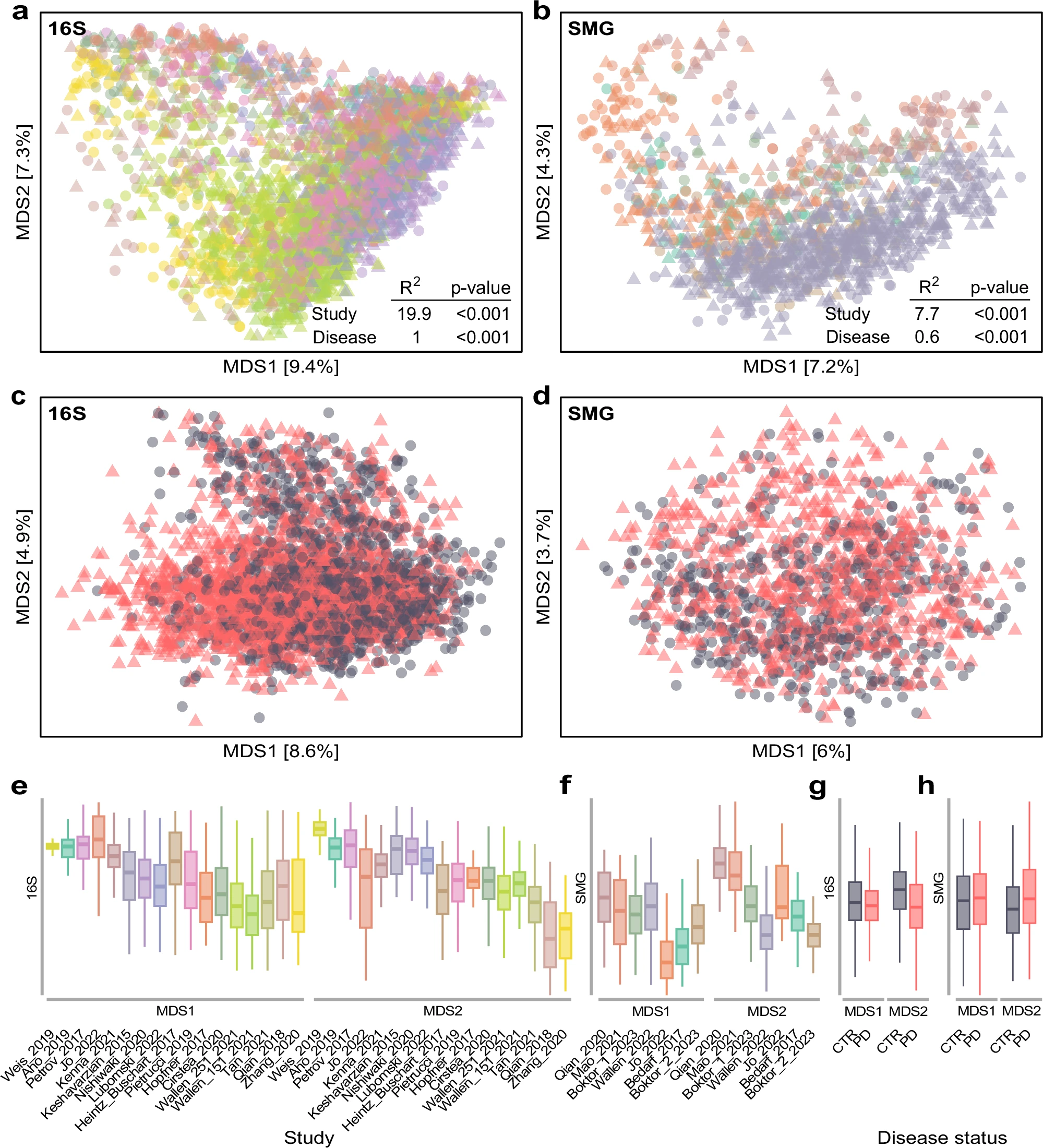
帕金森疾病患者和对照个体机体的肠道微生物组组成明显不同
这项研究中,研究人员纳入了来自11个国家、4个大洲的22项帕金森病与对照组的病例对照研究,共4489个样本,他们对这些样本进行了16S rRNA基因扩增子测序(16S)和宏基因组测序(SMG)分析。研究人员还使用了DADA2算法处理16S数据,通过mOTUs v3.0进行宏基因组数据的分类分析,并利用gffquant工具结合GMGC人类肠道基因目录进行功能分析。此外,他们还采用了包括随机森林、岭回归和LASSO回归等多种机器学习算法来评估肠道微生物组特征在帕金森疾病诊断中的准确性。
首先,研究人员对所有样本的肠道微生物组数据进行了标准化处理和质量控制,随后通过距离冗余分析(dbRDA)和置换多变量方差分析(PERMANOVA)评估样本的微生物组结构差异,同时他们还利用机器学习模型对每个数据集单独进行训练和验证,评估模型在区分帕金森病患者和对照组方面的表现,研究者还进行了跨研究验证(CSV)和留一研究验证(LOSO)分析来评估模型的泛化能力。
研究结果表明,在大多数研究中,基于肠道微生物组的机器学习模型能准确区分帕金森疾病患者和对照组,平均AUC值为71.9%。然而,这些模型大多具有研究特异性,其难以在其它研究中泛化应用,平均AUC值仅为61%。通过在多个数据集上训练模型就能显著提高模型的泛化能力和疾病特异性,平均LOSO AUC值达到68%。此外,研究人员还发现,帕金森疾病患者的肠道微生物组中与短链脂肪酸(SCFA)产生相关的细菌减少,而与炎症和肠道健康不良相关的细菌水平也会增加,这些发现为帕金森疾病的早期诊断和治疗提供了新的视角。
综上,本文通过大规模的元分析揭示了帕金森病患者肠道微生物组的显著变化,并评估了其在疾病诊断中的潜在应用价值,研究人员通过整合多个数据集提高了机器学习模型的泛化能力和疾病特异性,为未来基于肠道微生物组的诊断工具开发提供了重要参考,未来他们将会进行更大的多中心研究来进一步验证这些发现,并探索肠道微生物组与帕金森病之间的因果关系。(100yiyao.com)
参考文献:
Romano, S., Wirbel, J., Ansorge, R.et al..Nat Commun16, 4227 (2025). doi:10.1038/s41467-025-56829-3
版权声明 本网站所有注明“来源:100医药网”或“来源:bioon”的文字、图片和音视频资料,版权均属于100医药网网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:100医药网”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。 87%用户都在用100医药网APP 随时阅读、评论、分享交流 请扫描二维码下载->
