Nat Genet:黄海亮/葛天合作开发跨人群精细映射(fine
时间:2024-09-02

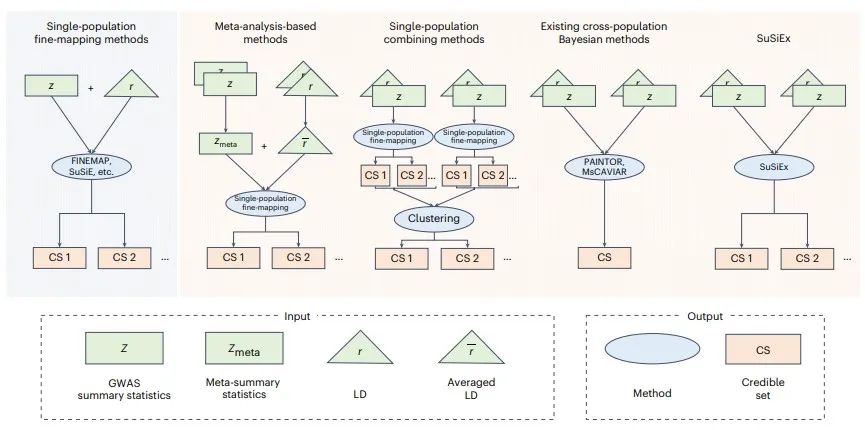
精细映射分析一览(Credit: Nature Genetics)
研究者首先进行了一系列全面的模拟,系统评估了SuSiEx的性能。通过改变群体间遗传相关性、因果突变数量、遗传力等模拟参数,从多个角度(如检测效力、覆盖度、准确度、分辨率、鲁棒性等)对SuSiEx进行了评估。模拟结果表明,SuSiEx在覆盖度、准确度和鲁棒性方面表现出色,并且相比单群体精细映射方法,SuSiEx具有更高的检测效力和分辨率。接下来,研究者将SuSiEx与其他现有方法(如单群体精细映射合并分析方法、基于元分析的精细映射方法、贝叶斯跨群体精细映射分析方法)进行比较,结果显示SuSiEx在易用性、分辨率和准确度等方面表现更佳。
随后,研究者将SuSiEx应用于不同人群生物样本库的数据分析。具体而言,他们将该方法应用于英国生物银行(UK Biobank)的欧洲人和非洲人群体数据,以及台湾生物银行(Taiwan Biobank)的东亚人群体数据。研究者筛选出25个在这两个数据库中共有的数量性状,并选出13,420个可能包含因果变异的基因组区域。通过使用SuSiEx分析三个人群的数据,研究人员共发现了14,361个99%置信集合(credible set),即该集合有99%概率包含因果突变。与仅使用单一人群数据的精细映射分析相比,SuSiEx在跨人群数据分析中发现了更多置信集合,其最佳位点的后验包含概率(Posterior Inclusion Probability, PIP)也更高,且后验包含概率大于95%的置信集合数量也显著增加。这些结果都凸显了SuSiEx方法及其在跨人群精细映射分析中的优越性。
最后,研究人员利用SuSiEx对精神分裂症进行了精细映射研究。他们使用了 精神类疾病基因组学研究联盟(Psychiatric Genomics Consortium, PGC) 提供的欧洲人和东亚人群体的精神分裂症GWAS汇总统计量和连锁不平衡信息【5】。SuSiEx在193个基因组关联区域中成功识别出215个99%置信度的置信集合,其中213个被推断为欧洲人群中的因果突变,95个被推断为东亚人群中的因果突变。在这213个置信集合中,有11个包含后验包含概率大于95%的突变位点。与预期一致,SuSiEx的表现优于已发布的PGC精细映射分析结果。
总体而言,文章介绍了一种精准高效的跨人群精细映射分析工具SuSiEx。通过整合多人群数据、明确建模群体特异性等位基因频率和连锁不平衡模式,SuSiEx发现了基因组中的更多因果变体,并可使用GWAS汇总统计量进行分析。
