Cell Res:左二伟团队开发AI模型——AlphaCD,高精度表征胞嘧啶脱氨酶
时间:2025-08-19
来源:生物世界 2025-08-19 10:32
该研究构建了一个机器学习模型——AlphaCD,该模型在预测胞嘧啶脱氨酶(CD)的催化效率、脱靶活性、靶位点窗口和催化基序方面均表现出高精度。基因组学的迅速发展使众多新蛋白质得以探索,这为表征生物体中蛋白质的功能带来了前所未有的机遇和新的挑战。尽管传统的生化实验方法在研究蛋白质功能方面发挥了重要作用,但这些方法存在明显的局限性,因为它们通常耗时费力,而且难以扩大规模以实现高效筛选。
蛋白质的一级序列(即氨基酸序列)对于预测蛋白质的功能至关重要,蛋白质序列分析能够识别功能基序和保守结构域,而序列比对则能揭示出可能具有功能参考价值的同源蛋白质。AlphaFold2的发展使得基于结构(而非序列)同源性的聚类方法得以应用,从而能够将大量可能具有相似功能的蛋白质进行分组。然而,这些方法无法对复杂且可能具有多种功能的蛋白质进行全面评估。
因此,迫切需要高精度、定量的方法来高效表征蛋白质的功能,以克服当前精度较低、定性的方法所带来的障碍。
2025 年 8 月 18 日,中国农业科学院深圳农业基因组研究所左二伟团队在Cell Research期刊发表了题为:AlphaCD: a machine learning model capable of highly accurate characterization for 21,335 cytidine deaminases的研究论文。
该研究构建了一个机器学习模型 AlphaCD,该模型在预测胞嘧啶脱氨酶(CD)的催化效率、脱靶活性、靶位点窗口和催化基序方面均表现出高精度,并利用预测结构构建了一个兼具超高保真度和高效率的胞嘧啶碱基编辑器(CBE)。

序列数据库涵盖范围广泛但支持性证据有限,这阻碍了具有特定功能蛋白质的鉴定。在这项最新研究中,研究团队在 HEK293T 细胞中实验表征了 1100 个与 nCas9 融合的APOBEC 样家族胞嘧啶脱氨酶(CD)的催化效率、靶位点窗口、基序偏好和脱靶活性,从而生成了迄今为止最大的单个蛋白家族的实验验证功能数据集。
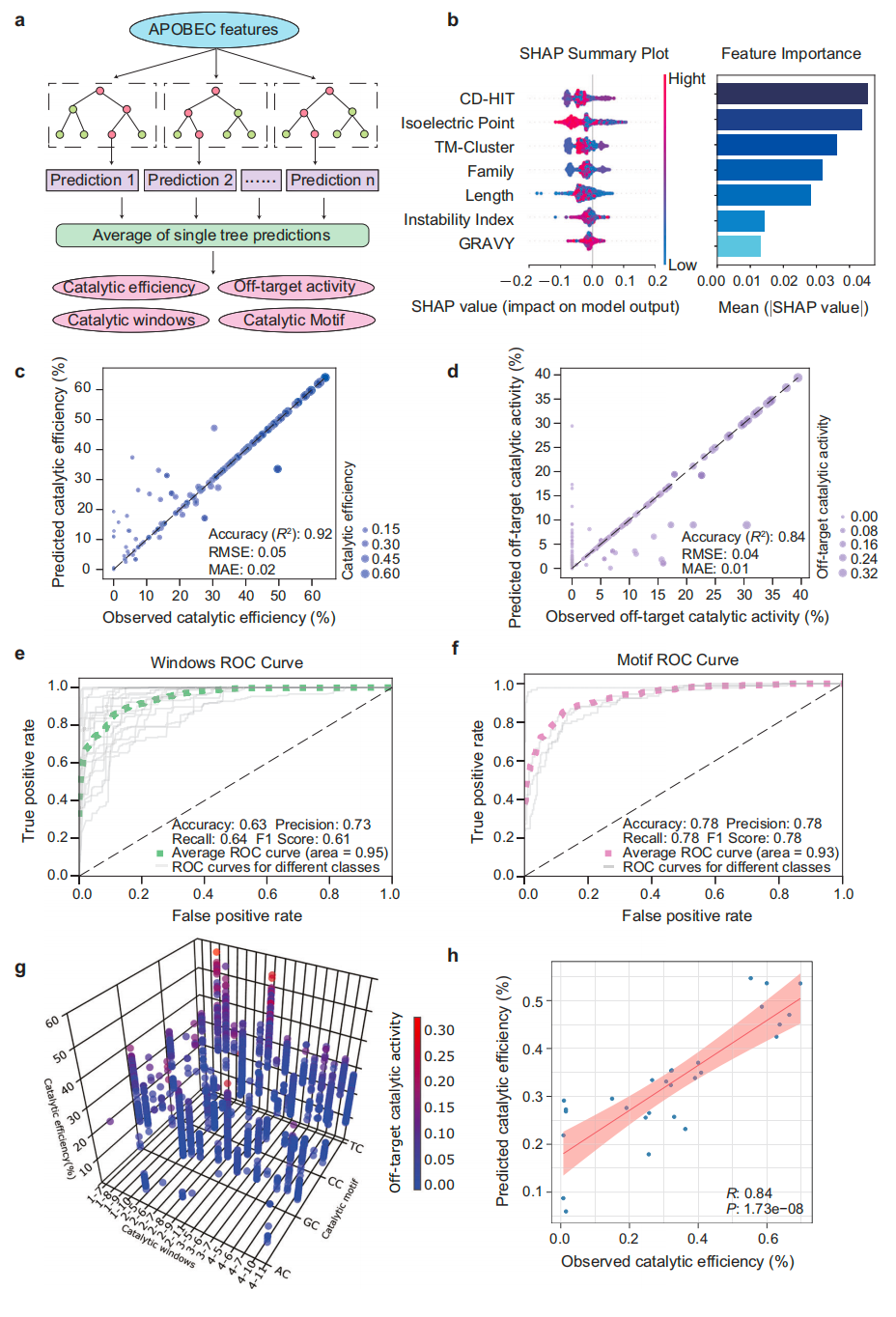
基于这些数据,结合氨基酸序列、三维蛋白质结构和 8 个附加特征,研究团队构建了机器学习模型 AlphaCD。该模型在预测催化效率(0.92)、脱靶活性(0.84)、靶位点窗口(0.73)和催化基序(0.78)方面均表现出高精度。研究团队将训练后的模型应用于预测 Uniprot 数据库中 21335 个胞嘧啶脱氨酶(CD)的上述催化特征,并对其中 28 个胞嘧啶脱氨酶进行子抽样验证,其预测精度分别为 0.84、0.87、0.75 和 0.73。
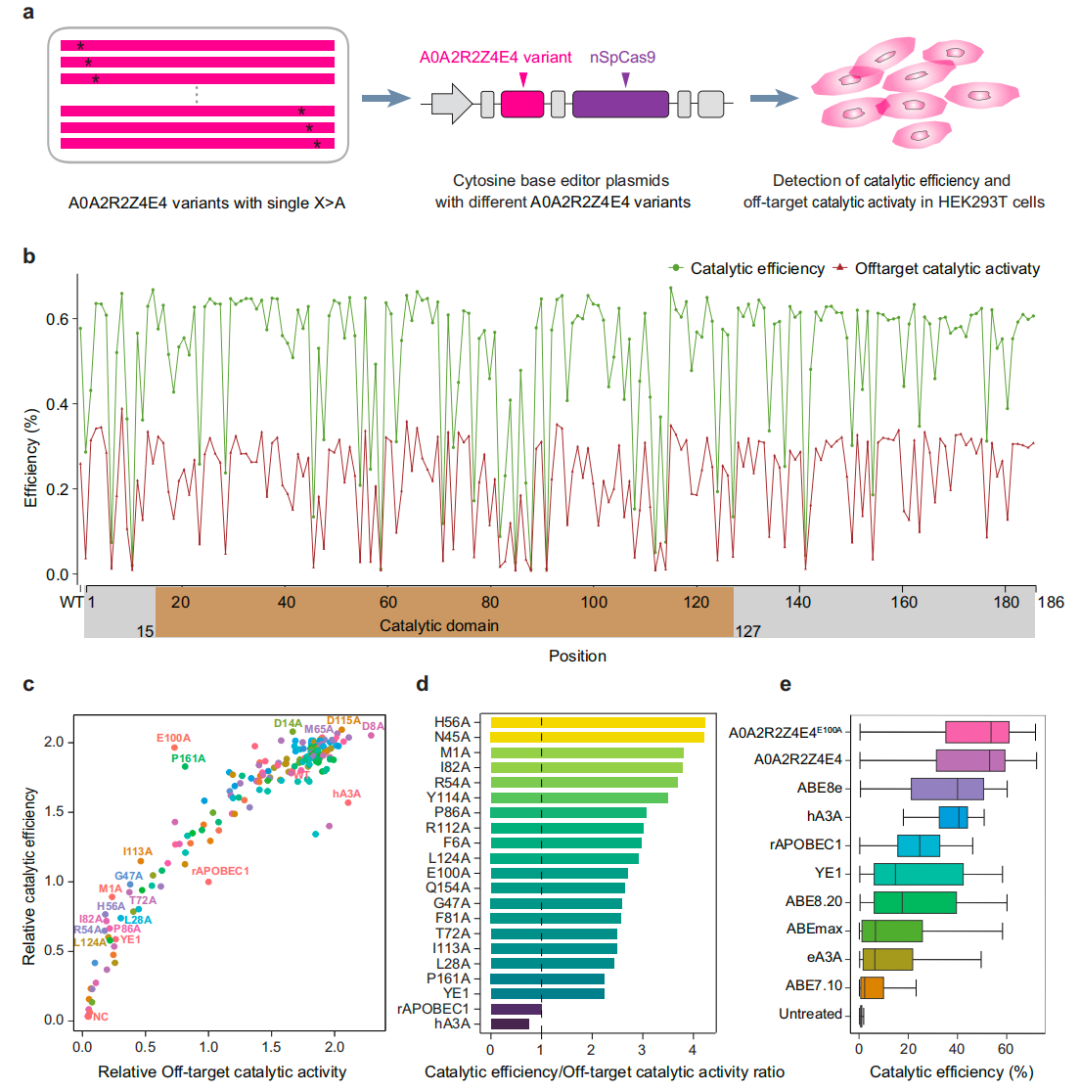
最后,研究团队通过基于丙氨酸扫描的诱变技术对其中一个胞嘧啶脱氨酶A0A2R2Z4E4的脱靶位点进行优化,构建了 A0A2R2Z4E4E100A变体,进而构建了兼具超高保真度和高效率的胞嘧啶碱基编辑器(CBE)。该案例不仅证明了 AlphaCD 在高精度、高通量蛋白质功能表征中的应用价值,还为其他蛋白质的加速功能解析提供了策略范式。